关于公共数据库
为了充分利用公开的SRA数据,实现最大化的利用数据,多维度、多角度的对公开的SRA数据进行深度发掘,我们整合了NCBI的所有的SRA公共数据。
高通量数据库的使用指南
名词解释
针对数据库可能出现的名词,做一下解释:
SRP:项目编号;
SRS:样品编号;
SRX:实验编号;
GSE:项目编号,即一个实验项目中的多个芯片实验,可能使用多个平台,可能涉及多个样本;
GSM:样品编号,即单个样本的实验数据;
原始测序数据(SRR)。
高通量数据库介绍
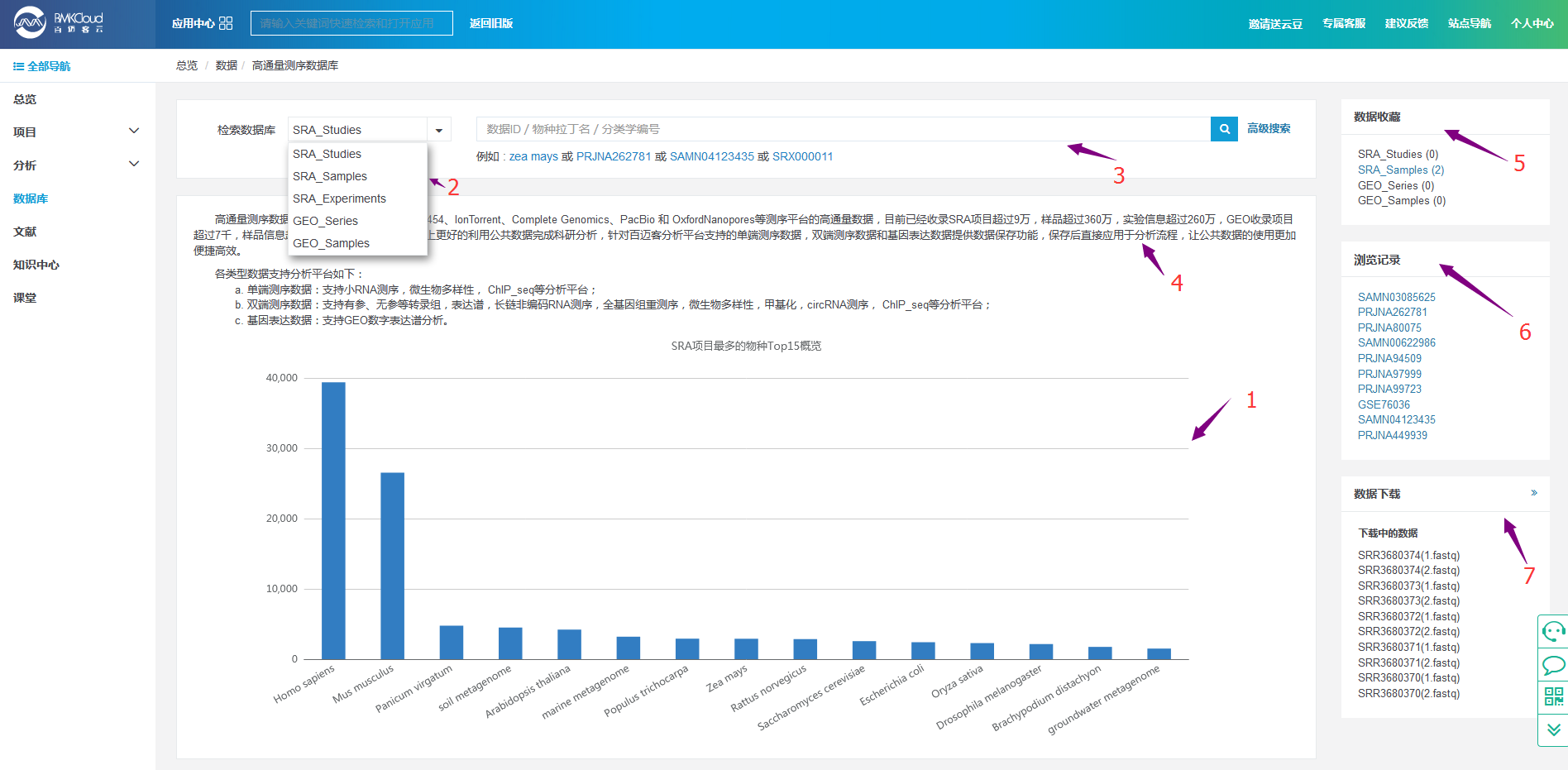
点击左侧导航的“数据库”,在数据库主页面中点击“高通量数据库”,即可进入此SRA数据库中进行查阅、检索:
标号“1”,以柱状图的形式统计出SRA项目最多的前15个物种的信息,点击默认的是,以特定的“物种”在SRA数据库中进行检索。
标号“2”,下拉框中说明“高通量数据库”中的5个子库分别是哪些:SRA的项目、SRA的样品、SRA的实验、GEO的项目、GEO的样品。
标号“3”,确定要在哪个子库中进行搜索框后,在搜索框中输入物种名、项目ID、实验ID或样品ID进行检索即可。
标号“4”,描述了目前SRA数据库中收录的数据信息。
标号“5”,“数据收藏”即为曾经在“高通量数据库”中进行收藏的数据的统计。
标号“6”,“浏览记录”即在此数据库中曾经做过的检索,保留了最新的10条检索记录。
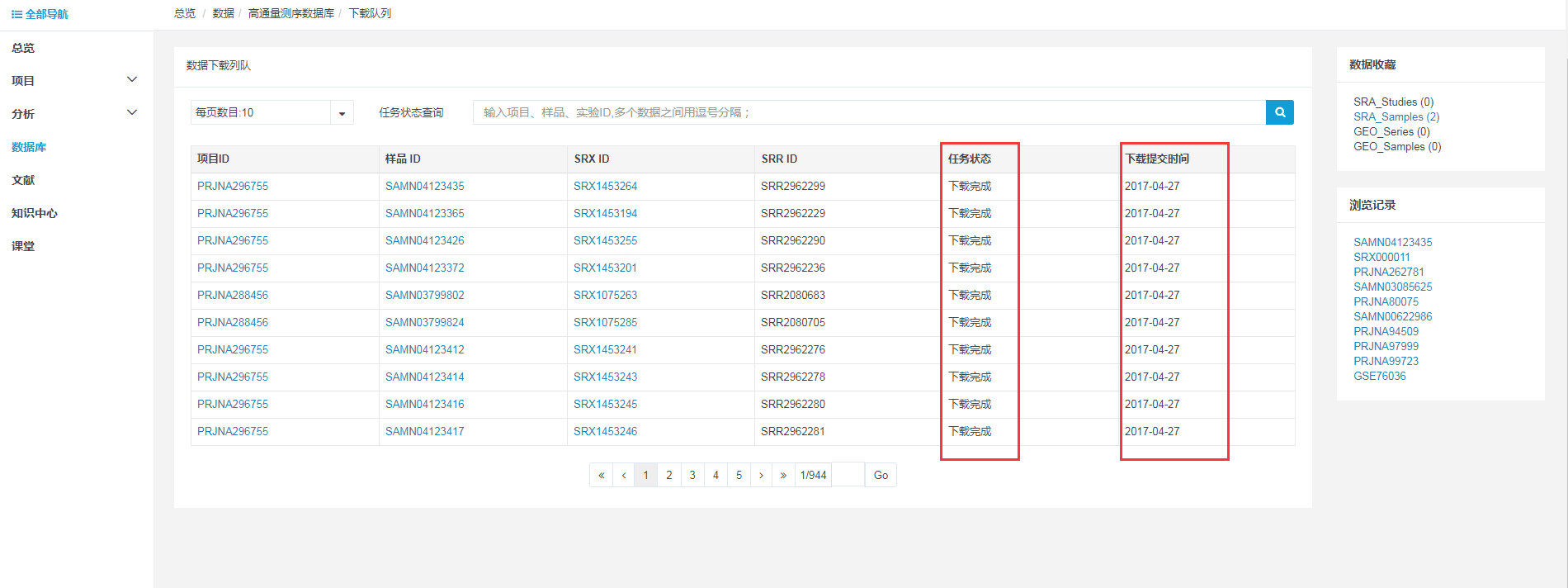
标号“7”,“数据下载”即平台上所有的下载数据的进度情况(包括他人的,方便了解目前平台下载情况),如下图:
可以查看目前数据下载的具体情况,如“任务状态”、“下载提交时间”等。
注:SRA_Studies:项目信息;SRA_Samples:样品信息;SRA_Experiments:实验信息;GEO_Series:GEO项目信息;GEO_Samples:GEO样品信息
数据库检索结果解读
下面以物种“zea mays”作为检索关键字进行结果的解释:
3.1 以物种作为检索关键字的结果页
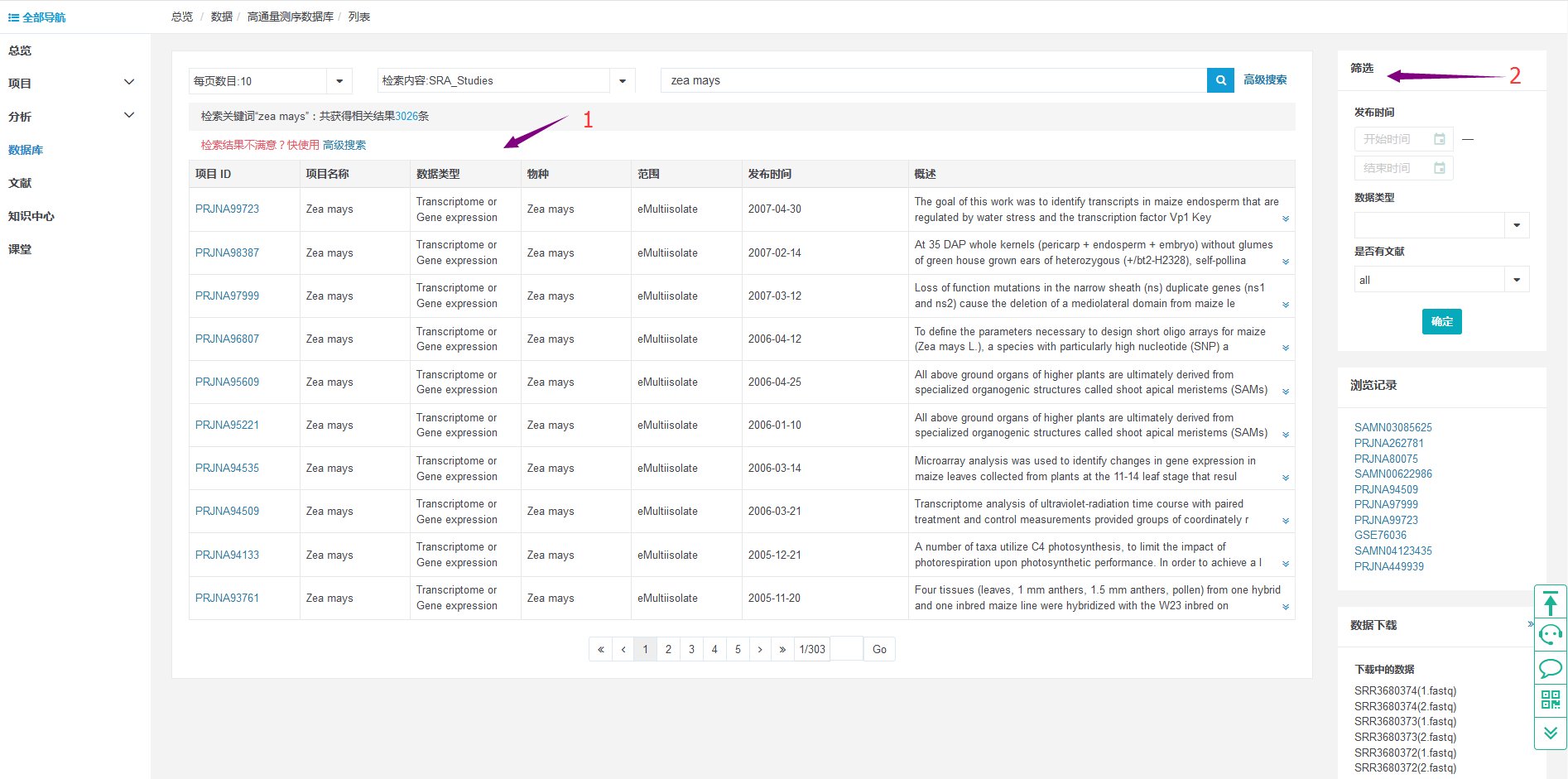
标号“1”,在子库“SRA studies”中,以“zea mays”检索的结果列表:
“项目ID”,出现此物种的所有的SRA项目ID;
“数据类型”,特定项目中的测试数据的类型;
“发布时间”,特定项目数据公布的时间;
“概述”,特定项目的目的、总结。
标号“2”,可以对检索得到的结果列表,做进一步的帅选:
条件1:“发布时间”,可以筛选,特定的起始时间,终止时间范围内的数据的信息,eg:起始时间:2017-09-20 结束时间:2018-09-21
条件2:“数据类型”,点击下拉框,可以筛选特定的数据类型的数据,eg:assembly
条件3:“是否有文献”,点击下拉框,可以筛选出包含(yes)、不包含(no)或所有的(all)项目数据。
选择了3个条件中的任何一个,点击“确定”,即可以在总的结果列表中进行筛选了。
3.2 项目ID对应的详情页面
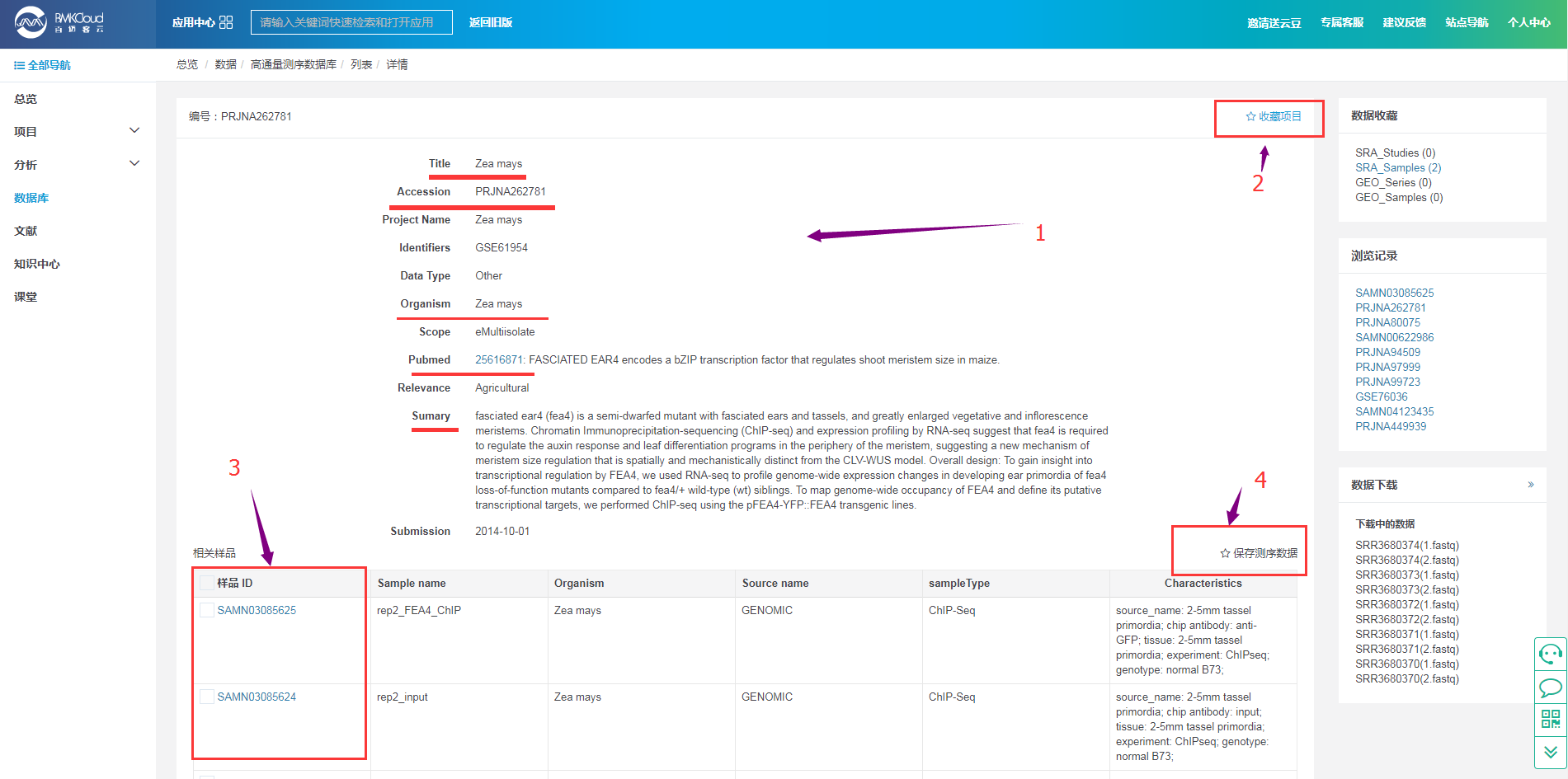
点击“项目ID”,即可得到此项目ID的详情页面:
标号“1”,是对整个项目的总体介绍。只有检索的关键字出现在“Title”或“Summary”中即可作为检索结果。
点击“Pubmed”,可以查看文献的详情信息。
标号“2”,如果此项目数据对自己有一定的价值,可以通过“收藏项目”见项目信息保存下来,便于后期查看。
标号“3”,此项目对应的所有的样品的信息,包括样品ID、样品的名称、物种来源、数据类型、测序类型、样品的一些简单的处理等描述信息;
标号“4”,选择有用的样品信息,点击“保存测序数据”即可以将数据下载下来,用于后期App分析等使用。
具体的下载进度,可以点击右侧的“数据下载”查看。
点击列表中的“样品ID”,可以得到样品的详情信息,见下文3.4。
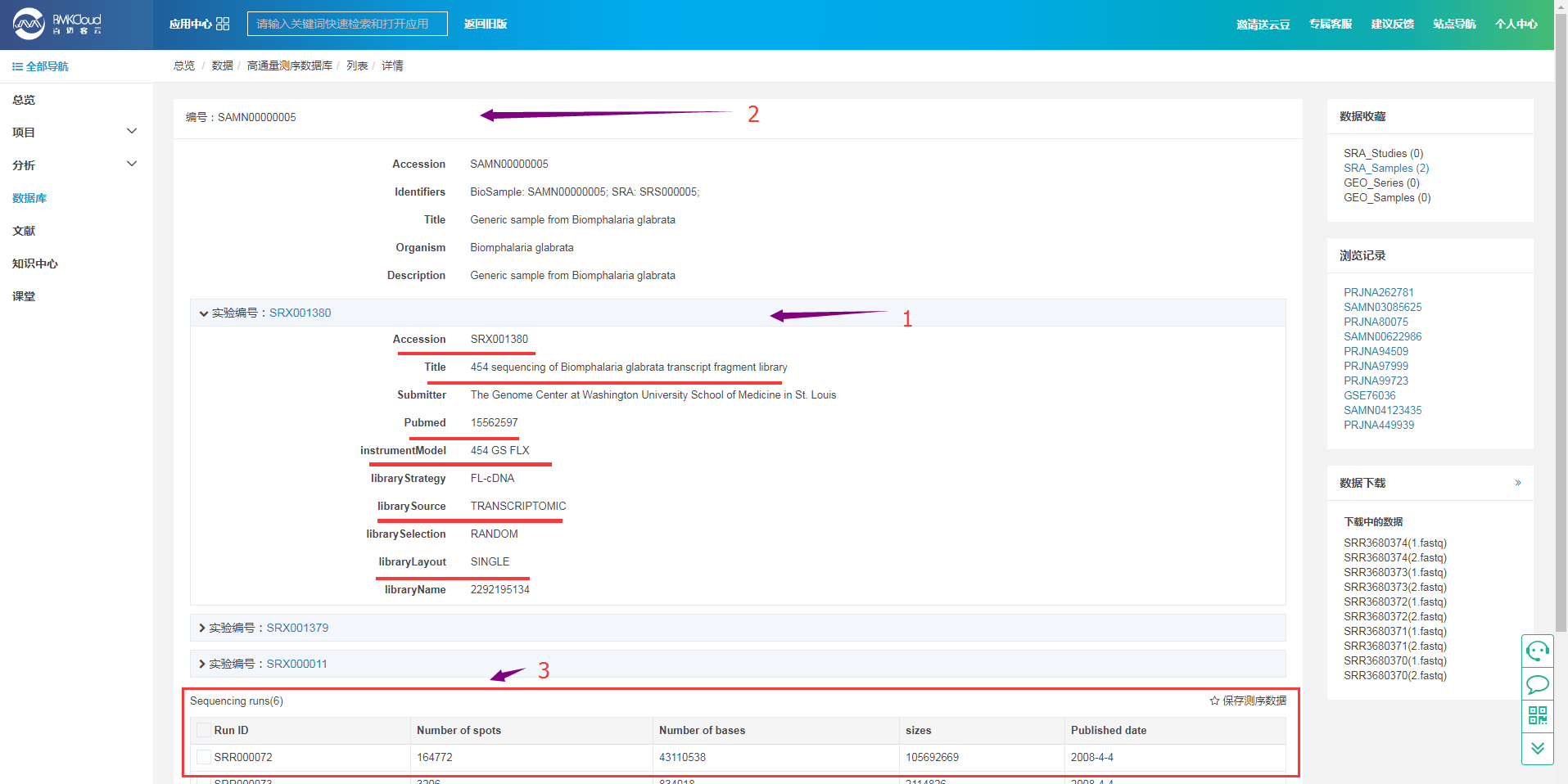
3.3 实验ID对应的详情页面
通过实验ID进行检索得到的详情页面:
标号“1”,是对检索的实验ID的详情描述信息,包括,实验对应的文献、此实验的测序平台、测序类型、单双端信息等等。
标号“2”,因为一个样品可能存在于不同的实验条件下,但是一个实验肯定属于一个样品,因此以样品ID为主导,将相关的实验关联在一起了。
因此在实验检索结果页面上,可能会看到多个实验ID的信息。
标号“3”,但是下面的测序Run信息是与特定的SRA 实验ID相对应的。 也就是说点击不同的SRA 实验ID 可得到与之对应的测序数据的信息。
点击“保存测序数据”,即可以进行数据的下载了。
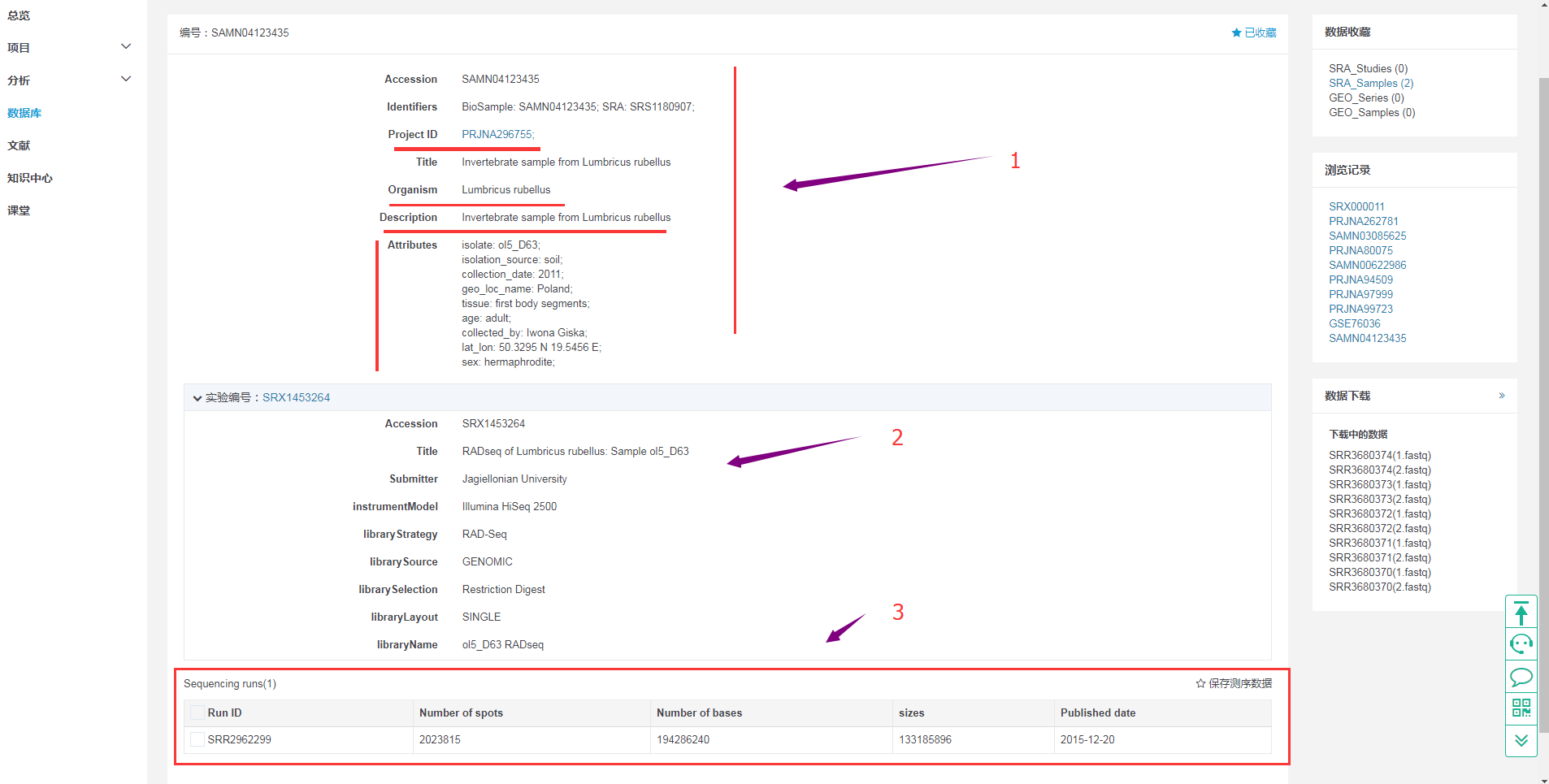
3.4 样品ID对应的详情页面
通过样品ID进行检索得到的详情页面:
标号“1”,是对应这个样品的描述信息,包括这个样品对应的项目编号、物种、这个样品对应的组织样的采集、处理等条件。
点击项目ID,可以查看项目的详情---对应页面解释如3.2
标号“2”,是对应的这个样品的实验的描述信息,默认的情况下,我们展开的是第一个实验ID的信息。
标号“3”,就是这个样品的测序情况。
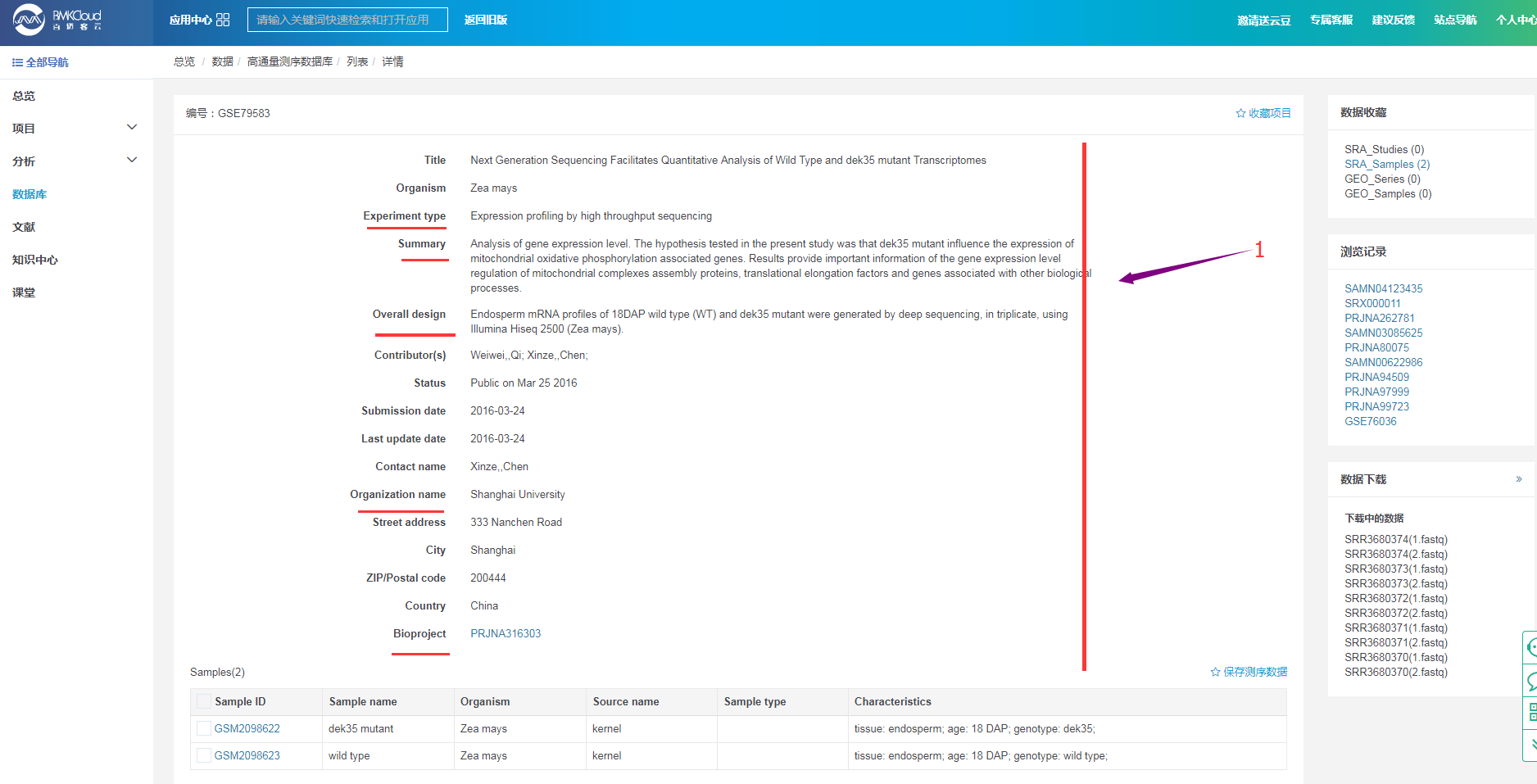
3.5 GEO项目ID对应的详情页面
通过GEO子库检索项目ID得到的详情页面:
除了此项目创建的背景的介绍等信息,还包括了此项目下对应的样品的信息。
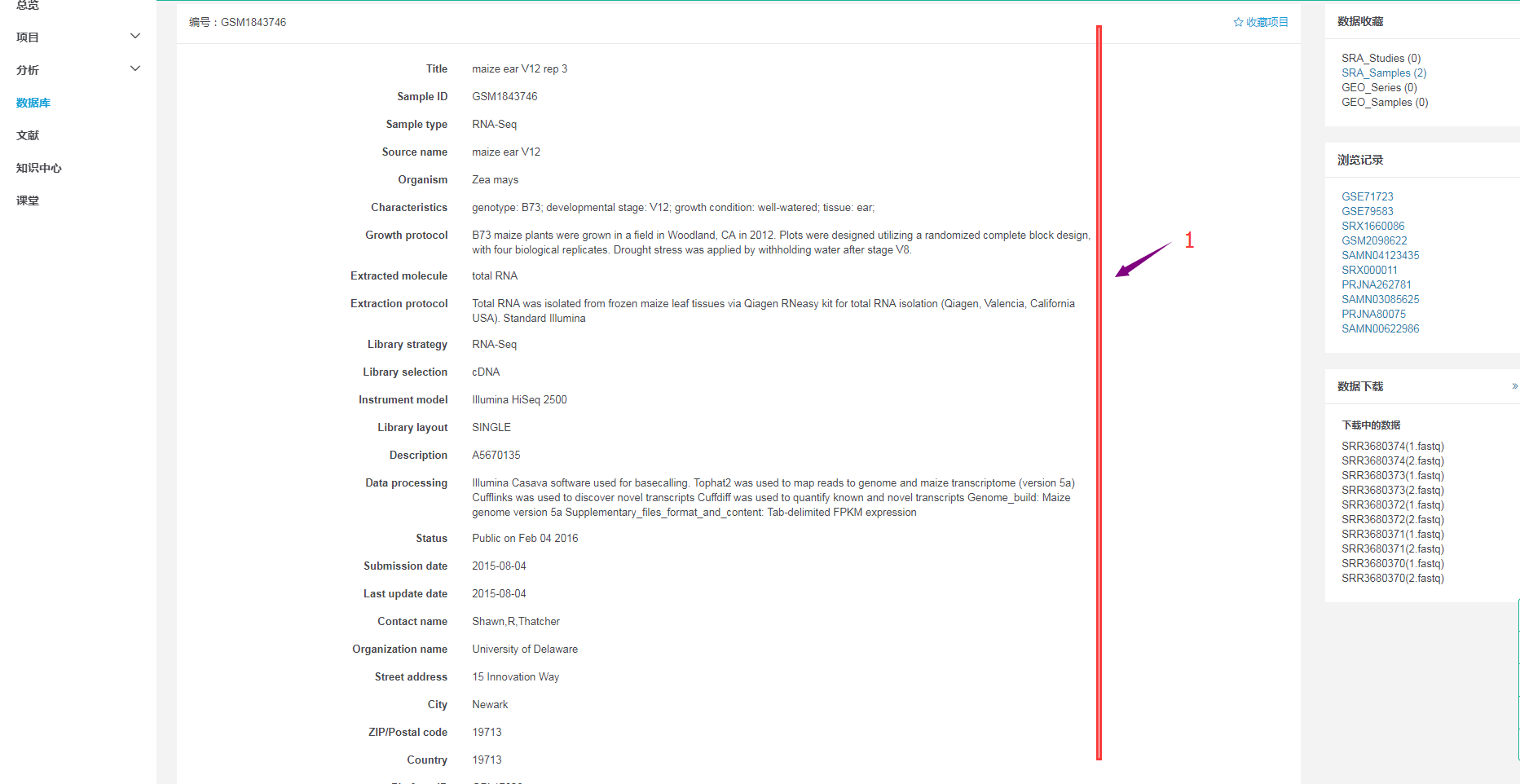
3.6 GEO样品ID对应的详情页面
通过GEO子库检索样品ID得到的详情页面:
包含了详情的样品的组织样收集信息,测序信息、提交者信息、提交日期更新日期等信息。
SRA和GEO数据库的优势
1.将SRA和GEO测序数据部署到云平台,使得用户更加快速、便捷的检索到特定物种的特定数据。
2.SRA和GEO数据库可以与云平台的APP想关联,利用公共数据结合APP进行数据深入挖掘。
应用场景
1.野生大豆泛基因组测序解密大豆遗传多样性及重要农艺性状基因:
中国农业科学院蔬菜花卉研究所自己测的7株野生大豆结合国外测的栽培性大豆,对野生大豆进行变异检测等进化研究等分析。
2.2016年在Biotechnology for Biofuels(IF 6.044)上再次发表了一篇文章《SorGSD: a sorghum genome SNP database》,构建了48个高粱品系总计62.9MSNPs的数据库SorGSD。
其中44个高粱是2014年由澳大利亚昆士兰大学、深圳华大等单位合作测的44个高粱重测序与2011年甜高粱和籽实高粱的全基因组遗传变异的数据结合构建的。