分析工具使用
工具一般是实现相对独立的功能,如绘制热图、绘制维恩图、序列比对、构建进化树等,针对比较个性化的分析内容,您可以通过串联几个工具来实现。相比分析平台,工具更灵活,但是工具不会生成完整的分析报告。
目前百迈客云平台包含3款工具集:
- 绘图工具:热图、维恩图、柱状图、饼图等
- FASTA文件工具集:提取、合并切分、转换、统计
- 表格工具:表格排序、筛选、合并
以及100+其他类型的工具,涵盖:绘图、质控、数据提取、序列分析、比对、统计、遗传进化、基因分析、组装、ncRNA等。
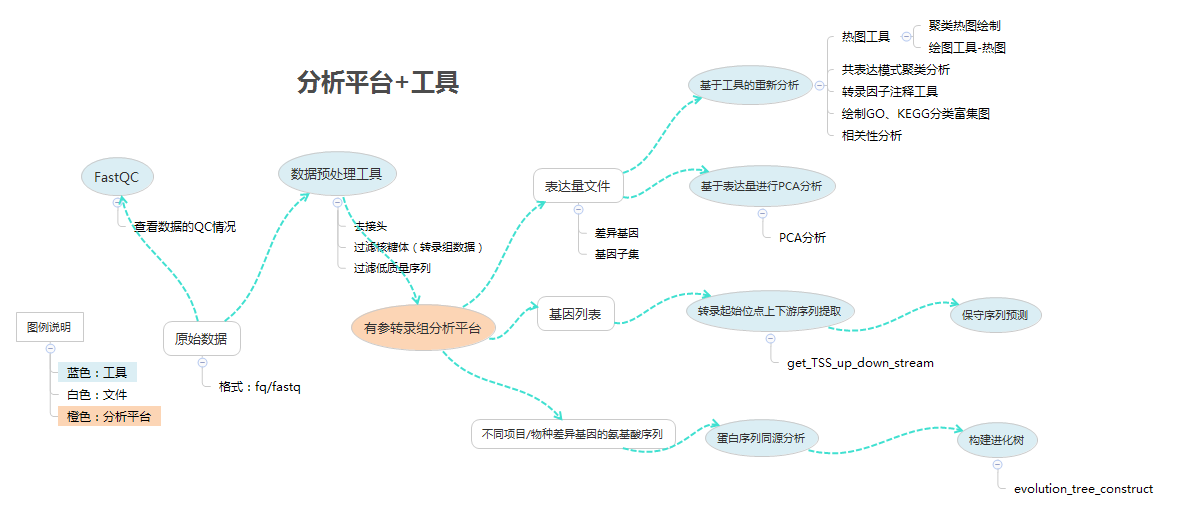
分析平台的分析结果可以用工具再次进行分析和绘图,下图中展示了有参转录组分析平台的分析结果可以关联的工具:
目前注册用户可以通过消耗云豆使用其中的70+款工具,另外30+款工具,由于消耗计算较多,因此暂时未对注册用户开发。所有工具及消耗云豆情况见下表:
./tools-on-bmkcloud/img/工具资源使用情况及云豆-201807.csv
| 工具中文名 | 工具英文名 | 分类 | 消耗云豆 |
|---|---|---|---|
| FLASH | FLASH | 组装 | 160 |
| Abyss中小基因组组装 | Assemble_by_Abyss | 组装 | - |
| FASTA序列转换成sqn | tbl2asn | 质控 | 10 |
| 去除read两端低质量碱基 | fastq_trim | 质控 | 40 |
| FastQC | FastQC | 质控 | 80 |
| 过滤冗余reads工具 | fastq_filter_by_duplication | 质控 | 100 |
| 过滤fastq接头序列 | fastq_filter_by_adapter | 质控 | 120 |
| 过滤rRNA序列 | fastq_filter_by_rRNA | 质控 | 120 |
| 原始数据污染程度预估工具 | NT_blast | 质控 | - |
| 按大小截取测序数据 | fastq_resize | 质控 | - |
| 数据预处理工具 | rawdata_preprocess | 质控 | - |
| MEGA | MEGA | 遗传进化 | 160 |
| 关联区域分析 | ED_Association_Analysis | 遗传进化 | 180 |
| 基于vcf文件统计欧式距离 | VCF_to_ED | 遗传进化 | 180 |
| 遗传共线性分析 | DrawAligmentRalationMap | 遗传进化 | 260 |
| 蛋白序列同源分析工具 | orthomcl | 遗传进化 | - |
| 构建进化树 | evolution_tree_construct | 遗传进化 | - |
| 混池群体Fst分析 | popoolation2_fst | 遗传进化 | - |
| 基于structure进行群体结构分析 | structure_analysis | 遗传进化 | - |
| 贝叶斯方法构建进化树 | MrBayes | 遗传进化 | - |
| 基于admixture进行群体结构分析 | admixture | 遗传进化 | - |
| 根据ID列表提取fasta序列 | extract_fasta_seq_by_id | 序列分析 | 6 |
| 简单重复序列分析 | SSR_Analysis | 序列分析 | 12 |
| 密码子偏好性分析 | Codon_bias | 序列分析 | 45 |
| ORF提取与统计 | Orf_Extraction | 序列分析 | 50 |
| 提取CDS和Intron序列 | extract_fa_by_gff | 序列分析 | 60 |
| 成对fastq转fasta | 2fq_to_1fa | 序列分析 | 80 |
| 外显子与内含子预测工具 | Exon_intron | 序列分析 | 80 |
| FASTA文件工具集 | Fasta | 序列分析 | ‘2-10 |
| 信号肽预测 | SignalP_analysis | 序列分析 | - |
| 编码区与非编码区预测工具 | CDS_UTR_predict | 序列分析 | - |
| 转录因子注释工具 | Plant-TF | 序列分析 | - |
| 保守序列预测 | motif_discovery | 序列分析 | - |
| NESmapper核输出信号预测 | NESmapper | 序列分析 | - |
| NLStradamus核定位信号预测 | NLStradamus | 序列分析 | - |
| 样品间差异SNP/INDEL提取 | DiffSnpIndelExtract | 突变 | 8 |
| 过滤亲本SNP位点 | SNPSelectionFromFM | 突变 | 20 |
| SNP位点引物设计 | snp_primer | 突变 | 60 |
| GATK突变检测 | GATK_VariantCalling | 突变 | - |
| Annovar变异注释 | Annovar_Annotate | 突变 | - |
| CNVnator CNV检测 | CNVnator | 突变 | - |
| SLAF分析 | slaf_call_snp | 突变 | - |
| 无参SLAF的Fst和群体分析 | slaf_noref_fst_population_analysis | 突变 | - |
| 有参SLAF分析 | ref_slaf_call_snp | 突变 | - |
| BreakDancer SV检测 | breakdancer | 突变 | - |
| PCA分析 | PCA_analysis | 统计 | 8 |
| 相关性分析 | correlation_analysis | 统计 | 8 |
| 序列gap区统计 | get_fa_gap | 统计 | 20 |
| 统计比对上序列覆盖度 | get_coverage_from_alignment | 统计 | 80 |
| 特定碱基位点reads覆盖率统计 | Reads_Distribution_stat | 统计 | 200 |
| 按列提取信息 | cut_columns | 数据提取 | 5 |
| 基于hapmap文件提取SNP信息 | hapmap_to_snp | 数据提取 | 5 |
| 提取相应ID行信息 | data_extract_by_ids | 数据提取 | 8 |
| 提取位点上下游定长序列 | Seq_extract_by_len | 数据提取 | 10 |
| get_TSS_up_down_stream | get_TSS_up_down_stream | 数据提取 | 10 |
| 基于基因ID过滤GFF信息 | get_gene_position_by_gene_id | 数据提取 | 80 |
| 生物学重复对应的基因表达量取均值 | Combine_columns_and_get_average | 基因分析 | 2 |
| 共表达模式聚类分析 | coexpression_cluster | 基因分析 | 8 |
| 蛋白基因家族分类 | Pfam_scan | 基因分析 | 18 |
| 基因表达量T检验 | T-test_geneExpression | 基因分析 | 25 |
| 差异表达基因分析 | DEG_Analysis | 基因分析 | 100 |
| topGO基因功能注释 | topGO | 基因分析 | 120 |
| edgeR分析工具 | edgeR_Analysis | 基因分析 | 120 |
| CPC编码能力预测 | CPC | 基因分析 | 280 |
| CNCI编码能力预测 | CNCI | 基因分析 | 280 |
| 蛋白跨膜区预测 | TMHMM_for_Transmembrane | 基因分析 | - |
| 基因功能注释 | Anno_function | 基因分析 | - |
| 加权基因共表达网络分析 | WGCNA | 基因分析 | - |
| 基于InterPro数据库的蛋白基因家族分类预测 | InterProScan | 基因分析 | - |
| 基于高通量数据获取内参基因软件 | internal_control | 基因分析 | - |
| 绘制饼图 | Draw_pie_pic | 绘图 | 4 |
| 聚类热图绘制 | heatmap_draw | 绘图 | 8 |
| 注释结果饼状统计 | Plot_Pie_anno | 绘图 | 8 |
| 共表达聚类热图 | expression_cluster_heatmap | 绘图 | 8 |
| 绘制GO、KEGG分类富集图 | draw_GO_KEGG_Graph | 绘图 | 10 |
| 绘制直方图 | facetGridBar | 绘图 | 10 |
| 简单柱状图 | simple_bar | 绘图 | 12 |
| 绘制差异基因MA图 | MA_plot | 绘图 | 12 |
| 绘制差异基因火山图 | Volcano_plot | 绘图 | 12 |
| 饱和度曲线图 | Saturation_plot | 绘图 | 12 |
| KEGG分类图 | KEGG_Classification_Plot | 绘图 | 15 |
| 绘制簇状柱状图 | dodged_bar | 绘图 | 15 |
| 绘制分组柱状图 | facet_wrap_bar | 绘图 | 15 |
| 分组数据点线图绘制 | multiData_point_line_draw | 绘图 | 18 |
| 多平面(纵向)点线图绘制 | horizontal_point_line_draw | 绘图 | 18 |
| 分组数据多平面点线图绘制 | facet_point_line_draw | 绘图 | 20 |
| 分组数据面积图绘制 | multiLevel_Area_draw | 绘图 | 20 |
| 差异基因GO富集分析柱状图 | GO_bar | 绘图 | 20 |
| 绘制KEGG各通路基因富集点状图 | KEGG_dot_plot | 绘图 | 25 |
| 单因素曼哈顿图 | manhattan_onefactor_plot | 绘图 | 28 |
| 染色体reads分布图 | Random_distribution_plot | 绘图 | 80 |
| 参考基因组reads统计分布图 | Reads_Distribution | 绘图 | 125 |
| Circos绘制 | draw_circos | 绘图 | 220 |
| 绘图工具 | Drawing | 绘图 | 0 |
| 表格文件转tab文件 | read_window_excel | 表格处理 | 2 |
| 分隔符转换 | delimiters_to_tab | 表格处理 | 2 |
| 行列转置 | Transpose | 表格处理 | 15 |
| 将根据软件要求编写的list文件,转换成svg格式文件 | distribution_draw | 表格处理 | 80 |
| 表格工具 | form_edit | 表格处理 | 0 |
| SAM/BAM互换 | SAMtools | 比对 | 120 |
| BLAT | BLAT | 比对 | - |
| BLAST | BLAST | 比对 | - |
| 蛋白质多序列比对工具 | Muscle | 比对 | - |
| 短序列比对工具bwa | bwa_aligner | 比对 | - |
| 多序列比对工具 | Clustal_omega | 比对 | - |
| 短序列比对 | SOAP_aligner | 比对 | - |
| 短序列比对工具bowtie2 | bowtie2_aligner | 比对 | - |
| lncRNA分类及对应基因注释 | lncRNA_toolkit | ncRNA | 8 |
| miRNA基因家族分类 | miRNA_family | ncRNA | 150 |
| miRNA序列ID统计 | miRNA_miRBase_alignment | ncRNA | - |
| microRNA靶基因预测软件 | TargetFinder | ncRNA | - |